SAM-Med3d三维医学图像通用分割大模型
应用概述
在医学影像领域,图像检测是一项至关重要的任务,它直接关联到早期诊断和治疗计划的制定,极大地影响着患者的治疗效果和生存率。传统的手工诊断方法已经无法满足快速、准确诊断的需求。随着深度学习技术的发展,基于人工智能的肿瘤图像检测方法成为研究热点。
使用场景
本应用可作为医学影像检测作为早期发现和诊断疾病的重要手段,对医学图像进行分析,旨在自动化地识别和定位图像中的病灶区域,从而为医生提供高效、准确的诊断依据。这一技术不仅可以显著提高病灶检测的效率和准确率,还能帮助医生评估疾病的进展情况和治疗效果,对提高患者的生存率和生活质量具有重要意义。
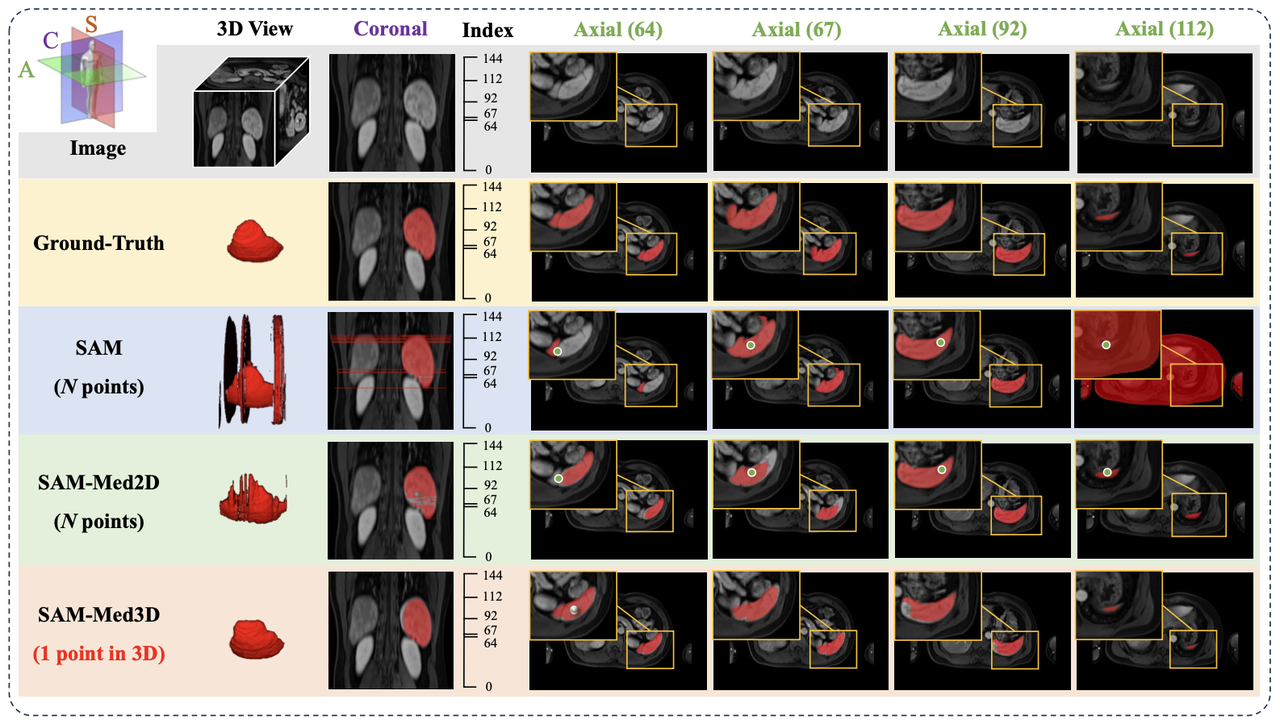
图一
图1:SAM 相关的模型在三维医学图像数据上的表现,SAM 和 SAM-Med2D 在空间上都出现了断层的现象,而 SAM-Med3D 在空间上具有更好的连贯性本应用经部署后,可以提供以下服务:
1.基于医学场景特定数据集进行模型微调
2.应用于医学领域特定场景的推理
操作步骤
部署前准备工作
Docker环境安装请参考官方文档:Docker环境安装教程
kubectl安装请参考官方文档:安装命令行工具(kubectl)
开通集群
资源最低要求
| 资源类型 | 数量 | 说明 |
|---|---|---|
| GPU | 1个起 | gpu-h800 |
| CPU | 16 core起 | |
| 内存 | 320G 起 | |
| 存储 | 2T 起 | |
| Harbor存储 | 100G | 用于保存镜像 |
| 对象存储 | 300G | 用于保存模型和数据 |
申请开通
集群请参考:开通弹性容器集群
集群开通后,需要将集群配置信息保存到合适的位置,以便后续使用。
脚本准备
请下载模板脚本文件,并解压到本地目录。
在前面下载并解压的模板脚本文件中,对所有文件进行变量替换。
需要替换的变量有:
| 变量名 | 所在文件 | 说明 | 来源 | 例子 |
|---|---|---|---|---|
| harbor_endpoint | harbor-config.json | 镜像仓库访问域名 | 登录网页-资源中心-存储管理-对象存储-访问详情页 | registry.hd-01.alayanew.com:8443 |
| harbor_username | harbor-config.json | 镜像仓库用户名 | 镜像仓库开通短信 | hb_abc123 |
| harbor_password | harbor-config.json | 镜像仓库密码 | 镜像仓库开通短信 | 123456 |
secret准备
secret文件是用于保存敏感信息的,如对象存储的密钥等。本次部署中需要对harbor registry的用户名和密码进行加密,并保存到secret文件中。
- 当执行过前面的步骤后,请将harbor-config.json中的内容做base64编码。linux中可以使用base64命令进行编码。或者在线网站也可以进行编码。可以参考:https://tool.lu/encdec/
- 将编码后的内容保存到harbor-secret.yaml的.dockerconfigjson字段。 例子:
apiVersion: v1
kind: Secret
metadata:
name: sam-med3d-secret
namespace: sam-med3d
type: kubernetes.io/dockerconfigjson
data:
.dockerconfigjson: <base64编码后的内容>
镜像准备
包括以下步骤:
1.编写Dockerfile文件
2.构建镜像
3.推送镜像
以下镜像仓库信息使用自己的
用户名密码:查看开通镜像仓库时的通知短信
镜像仓库访问地址:参考镜像仓库的使用
镜像仓库访问地址:是由 访问域名/项目 组成
关于如何查看镜像仓库,请参考:使用harbor管理镜像资源
1:创建Dockerfile
FROM swr.cn-north-4.myhuaweicloud.com/ddn-k8s/docker.io/library/ubuntu:22.04
ARG DEBIAN_FRONTEND=noninteractive
RUN sed -i 's|http://ports.ubuntu.com|https://mirrors.tuna.tsinghua.edu.cn|g' /etc/apt/sources.list
RUN apt update -y && \
apt install -y curl wget
ENV PATH /opt/conda/bin:$PATH
RUN . ~/.bashrc && \
/opt/conda/bin/conda init bash && \
conda info --envs
WORKDIR /
RUN mkdir ~/.pip
RUN echo "[global]" > ~/.pip/pip.conf
RUN echo "index-url = http://pypi.douban.com/simple" >> ~/.pip/pip.conf
RUN echo "[install]" >> ~/.pip/pip.conf
RUN echo "trusted-host=pypi.douban.com" >> ~/.pip/pip.conf
COPY rclone.sh .
RUN bash rclone.sh
RUN mkdir -p /root/.config/rclone/
EXPOSE 22
COPY requirements.txt /tmp/requirements.txt
#装载镜像所需的python包
RUN pip3 install -r /tmp/requirements.txt
#把项目文件拷贝到镜像
COPY . /mnt/
USER root
WORKDIR /mnt
2.构建镜像
#step2:构建镜像
sudo docker build . -t sam_med3d:1.0
3.推送镜像
登录镜像仓库,推送镜像。 当执行完毕后,请检查镜像仓库中是否有镜像文件。
#login
docker login [镜像仓库访问域名] -u [user] -p [password]
# tag
docker tag 镜像仓库访问地址/sam_med3d:1.0
# push
docker push 镜像仓库访问地址/sam_med3d:1.0
代码、模型、数据准备
模型文件:可根据具体场景需要选择特定场景的基模型
| Model | Google Drive | Baidu NetDisk |
|---|---|---|
| SAM-Med3D | Download | 百度网盘 |
| SAM-Med3D-organ | Download | 百度网盘 |
| SAM-Med3D-brain | Download | 百度网盘 |
| SAM-Med3D-turbo | Download | 百度网盘 |
数据文件:AMOS公共数据集 或准备自定义数据集,注意文件结构如下:
data/medical_preprocessed
├── adrenal
│ ├── ct_WORD
│ │ ├── imagesTr
│ │ │ ├── word_0025.nii.gz
│ │ │ ├── ...
│ │ ├── labelsTr
│ │ │ ├── word_0025.nii.gz
│ │ │ ├── ...
├── ...
代码文件:https://github.com/uni-medical/sam-med3d
数据文件较大(20G),请确保本地有足够的存储空间,由于文件较大,下载时间较长,请保证网络稳定。直接下载可能导致下载中断而需要重新下载。推荐使用下载工具下载。
准备工作包含以下步骤:
- 下载上面的模型文件和数据文件
- 上传到对象存储中。 对象存储的上传以及检查请参考:对象存储的使用
上传sam_med3d_turbo.pth模型文件到s3
rclone copy sam_med3d_turbo.pth [s3_store]:[/mnt/aiws/] --progress
上传已标注数据集到s3
rclone copy amos22 [s3_store]:[data_path] --progress
上传项目代码到s3
rclone copy [sam_med3d_pro] [s3_store]:[pro_path] --progress
部署应用
包含以下步骤:
- 执行k8s配置文件,部署应用
- 检查k8s部署结果
- 进入pod,并进行初始化
执行部署脚本
请在脚本目录下执行一下命令:
kubectl apply -k .
确保执行过程中没有错误。
检查部署结果
请执行以下命令:
kubectl get pods -n sam-med3d
如果出现以下输出,说明部署成功:
NAME READY STATUS RESTARTS AGE
sam-med3d-deploy1-6cff86cfc8-drb5d 1/1 Running 0 10m
其中sam-med3d-deploy1-6cff86cfc8-drb5d是pod的名字,STATUS为Running时pod创建成功。
进入pod,并进行初始化
1.将完整项目代码传至pod内
#获取pod
kubectl get pod -n sam-med3d
将完整项目代码传至pod内
kubectl cp [/mnt/aiws/services/SAM-Med3D-main] [pod]:/mnt/sam-med3 -n sam-med3d
#将项目文件放至镜像的工作目录
cp -r [./SAM-Med3D-main/*] [/mnt/sam-med3]
项目代码放至镜像的工作目录
2.进入pod中,从s3下载模型文件和训练数据
#进入pod
kubectl exec -it [pod] -n [namespace] -- bash
cd [workspace]
下载sam_med3d_turbo.pth模型文件到/[workspace]/ckpt/目录
mkdir ckpt
rclone -v copy s3_store:/mnt/aiws/sam_med3d_turbo.pth /[workspace]/ckpt/
#下载自定义微调数据集到/[workspace]/data/medical_preprocessed/目录
mkdir data
cd ./data
mkdir medical_preprocessed
rclone -v copy s3_store:[data_path] /[workspace]/data/medical_preprocessed/
开始使用
微调
进入/workspace/目录,执行以下命令
sh train.sh
推理
进入/workspace/目录,执行以下命令
sh infer.sh
评估
进入/workspace/目录,执行以下命令
sh val.sh
资源配置说明:
- cpu: 18
- memory: 200G
- gpu:nvidia.com/gpu-h800: 1
容器中的目录说明
- /workspace/ckpt:工作目录,存放项目代码、训练入口、配置文件等。
- /workspace/ckpt:模型目录,存放预训练的基模型。
- /workspace/data/medical_preprocessed:数据目录,存放已标注的训练数据。
关于license
请按照sam_med3d的版权要求使用,请参考:https://github.com/uni-medical/sam-med3d/LICENSE